Harmonizonme数据库
数据库概览
进入Harmonizonme主页(https://maayanlab.cloud/Harmonizome/),点击About可见数据库简介,于2016年发布,目前收录来自66个在线数据库的114个基因组学、转录组学、蛋白质组学和代谢组学数据集,着重于定义基因和不同属性之间的关联,属性可以是基因、蛋白质、细胞系、组织、实验干预因素、疾病、表型或药物,并构建基因与基因和属性与属性的相似性网络。本文重点介绍转录因子和靶基因数据集。
功能及操作演示
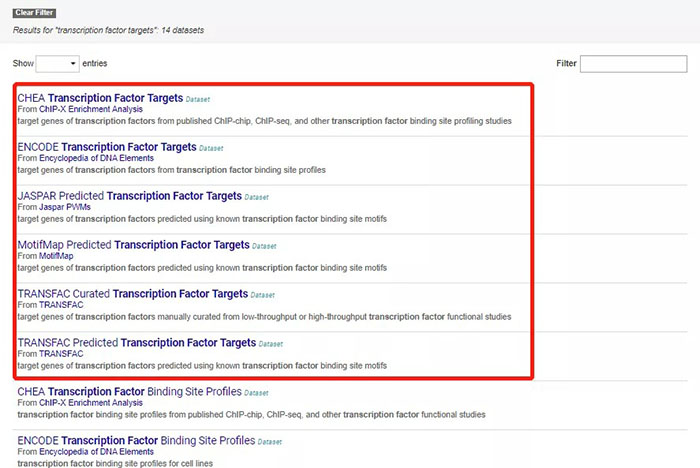
转录因子靶基因预测,进入Harmonizonme主页,点击SEARCH进入检索界面,下拉菜单选择Database,检索框输入transcription factors targets,回车得到14条检索结果,前6个为转录因子靶基因数据库。
选择CHEA Transcription Factor Targets进入功能页面,首先是该数据库简介,包括数据来源和参考文献,页面下拉依次是不同数据集下载链接,以及该数据库收录的各个转录因子详细信息。
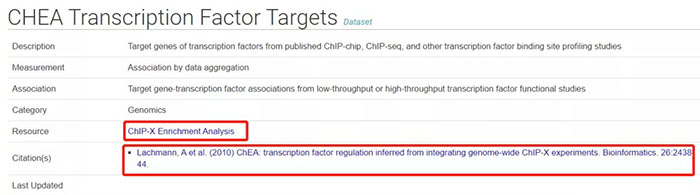

以人类SOX2为例,检索框输入SOX2,点击进入详情页面,页面下拉可见该数据库收录的SOX2靶基因共8156个。

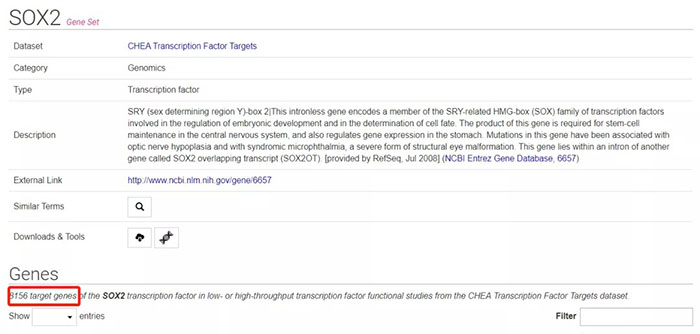
点击Downloads & Tools后面的下载按钮可以下载靶基因列表,该文件为Json文件,对于不熟悉R语言的小伙伴来说极为不友好,可按照另一种方法获取靶基因列表。

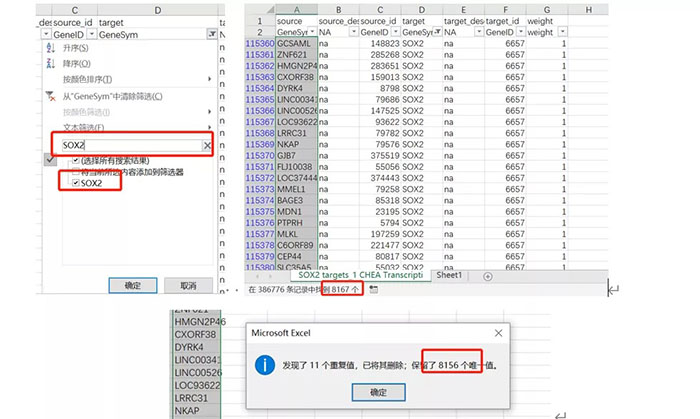
返回上一级页面,下拉到Data Access,点击 Gene-Attribute Edge List下载该数据库全部的转录因子和靶基因信息列表,用Excel打开,其中source列是靶基因,`target列是转录因子,分别提供Gene symbols和Gene ID。
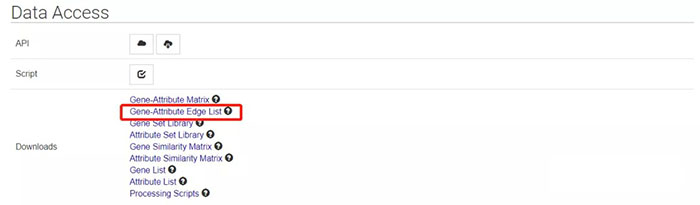
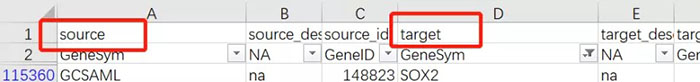
使用数据筛选功能,在target列检索SOX2,删除source列重复值,可以得到8156个靶基因结果。
同样的方法得到其他5个数据集收录的SOX2靶基因列表,JASPAR Predicted Transcription Factor Targets Dataset中有5个靶基因结果,TRANSFAC Predicted Transcription Factor Targets Dataset中有1228个靶基因结果,TRANSFAC Curated Transcription Factor Targets Dataset、MotifMap Predicted Transcription Factor Targets和ENCODE Transcription Factor Targets Dataset中无SOX2靶基因信息。
hTFtarget数据库
数据库概览
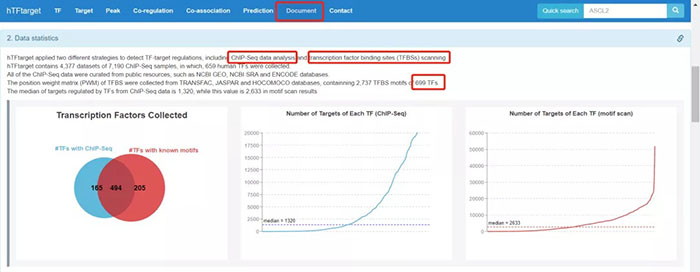
点击(http://bioinfo.life.hust.edu.cn/hTFtarget#!/),进入hTFtarget (Database of Human Transcription Factor Targets)主页,该数据库收录399种细胞系、129种组织或细胞和141种干预因素共569种条件下的7190个实验样本大规模ChIP-Seq数据中659个TFs相关信息。点击Document可看到hTFtarget中关于TFs靶基因数据是基于ChIP-Seq数据分析和TFBSs分析两方面的结果。
功能及操作演示
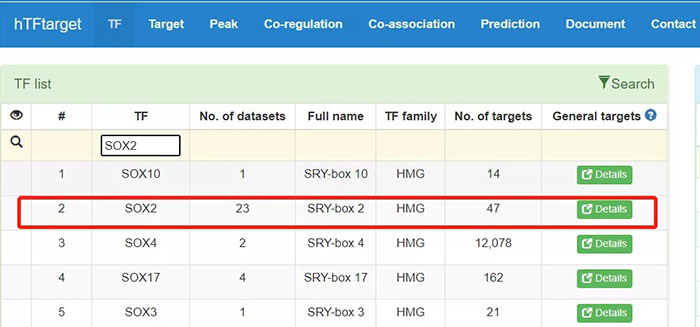
转录因子靶基因预测,进入hTFtarget主页,点击TF进入检索界面,检索框输入SOX2,点击进入详情页面, 点击下载按钮得到SOX2靶基因列表。
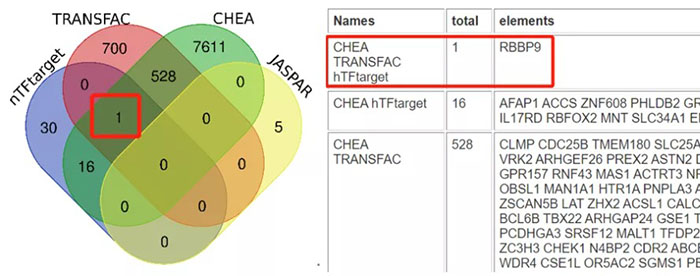
最后将以上两个数据库查到的靶基因列表绘制Venn图取交集,可以使用在线Venn图绘制工具(http://bioinformatics.psb.ugent.be/webtools/Venn/),分别输入CHEA中8156个靶基因,JASPAR中5个靶基因结果,TRANSFAC中1228个靶基因和hTFtarget获取的靶基因列表,结果显示4个数据库共有的SOX2靶基因数目为0,CHEA、TRANSFAC和hTFtarget共有的SOX2靶基因有1个,为RBBP9,Pubmed检索未见报道,可以尝试实验验证一波。
文献单图复现
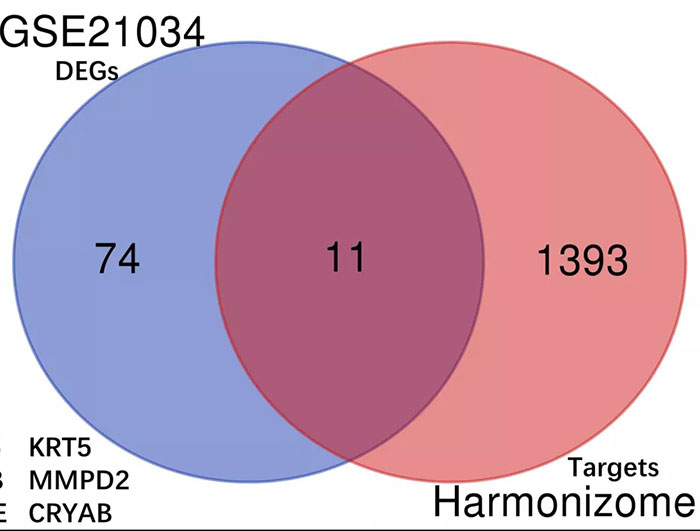
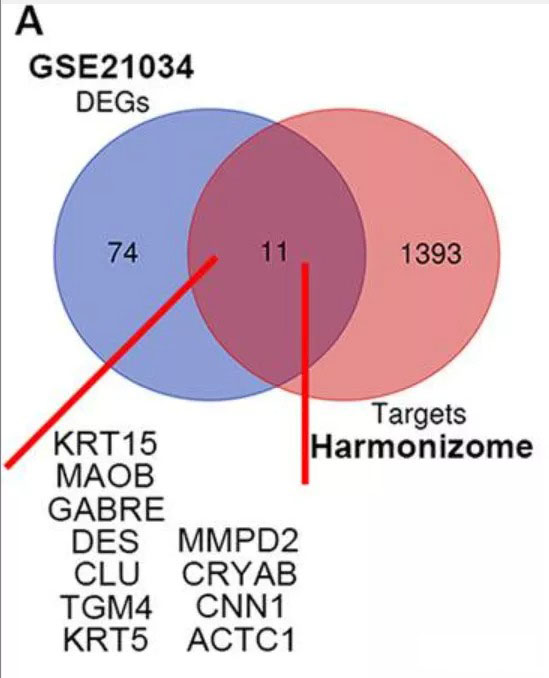
本文Figure6A,本文已实验验证主变量miR-671-5的靶基因为转录因子NFIA,随后通过Harmonizome预测到NFIA的1404个候选靶基因,并在GSE21034数据集获得85个表达差异基因(adjust_P<0.05,|Fold change | >2),二者取交集获得11个候选靶基因,再然后使用cBioPortal分析NFIA与11个候选靶基因相关性,结合Pubmed数据,以及qPCR和WB实验结果,最终能否证实NFIA可以靶向CRYAB并调节其表达。
单图复现如下:
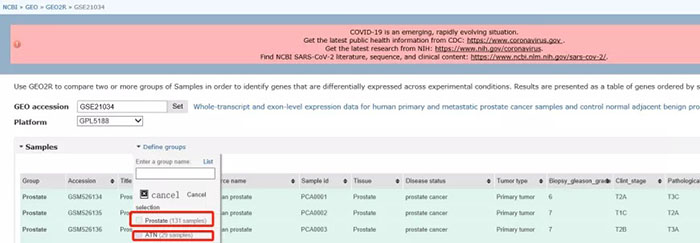
进入GEO数据库检索GSE21034,进入该数据集详情页面,可见该数据集包含GPL5188和GPL10264两个平台测序结果,点击Analyze with GEO2R,选择GPL5188平台加载185个样本,其中包含131例原发肿瘤组织和29例癌旁正常组织,分别添加到分组后,点击Analyze得到差异分析结果,下载后Excel打开,依次如下操作
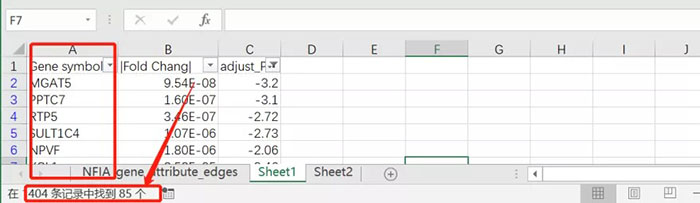
(1)一个基因对应多个探针的情况保留logFC最大值; (2)一个探针对应多个基因的情况删除该条目; (3)空白无名称条目删除; (4)筛选功能,筛选条件为adjust_P<0.05,|Fold change | >2。最终得到85个表达差异基因。
进入Harmonizonme主页,点击SEARCH进入检索界面,下拉菜单选择Database,检索框输入transcription factors targets,得到6个转录因子靶基因数据库,同前法查询NFIA靶基因,结果只有TRANSFAC Predicted Transcription Factor Targets中查询到NFIA的靶基因1404个,其余5个数据库未收录其靶基因信息,同前法获得NFIA的靶基因列表。
将GEO获得的差异表达基因与Harmonizonme获得的NFIA靶基因列表分别输入在线Venn图绘制工具(http://bioinformatics.psb.ugent.be/webtools/Venn/),即可得到Venn图,PS或AI添加交集部分包含的基因名称,即可得到本文Figure6A。